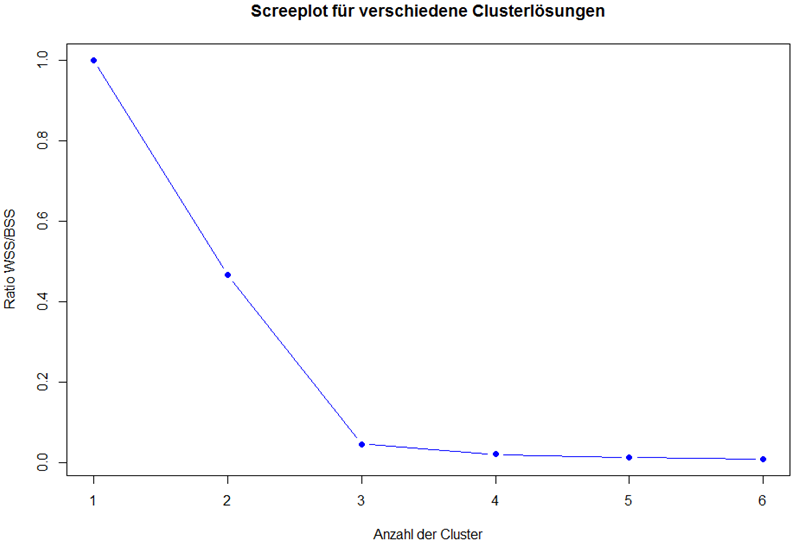
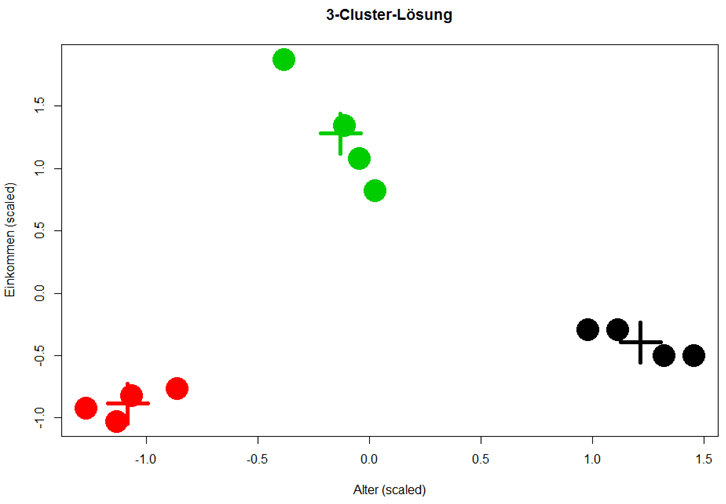
#R-script for emphasizing convergence and divergence of sample means
####install and load relevant packages ####
#uncomment these lines if necessary
#install.packages(c('ggplot2',’stabledist’))
#library(ggplot2)
#library(stabledist)
#####drawing random samples #####
#Setting a random seed for being able to reproduce results
set.seed(1234567)
N= 2000 #sample size
#Choose a PDF from which a sample shall be drawn
#To do so (un)comment the respective lines of following code
data <- rcauchy(N) # option1(default): standard Cauchy sampling
#data <- rnorm(N) #option2: standard Gaussian sampling
#data <- rexp(N) # option3: standard exponential sampling
#data <- rstable(N,alpha=1.5,beta=0) # option4: standard symmetric Holtsmark sampling
#data <- runif(N) #option5: standard uniform sample
#####descriptive statistics####
#preparations/declarations
SUM = vector()
sd =vector()
mean = vector()
SQ =vector()
SQUARES = vector()
median = vector()
mad =vector()
quantiles = data.frame()
sem =vector()
#piecewise calculaion of descrptive quantities
for (k in 1:length(data)){ #mainloop
SUM[k] <- sum(data[1:k]) # sum of sample
mean[k] <- mean(data[1:k]) # arithmetic mean
sd[k] <- sd(data[1:k]) # standard deviation
sem[k] <- sd[k]/(sqrt(k)) #standard error of the sample mean (for finite variances)
mad[k] <- mad(data[1:k],const=1) # median absolute deviation
for (j in 1:5){
qq <- quantile(data[1:k],na.rm = T)
quantiles[k,j] <- qq[j] #quantiles of sample
}
colnames(quantiles) <- c('min','Q1','median','Q3','max')
for (i in 1:length(data[1:k])){
SQUARES[i] <- data[i]*data[i]
}
SQ[k] <- sum(SQUARES[1:k]) #sum of squares of random sample
} #end of mainloop
#create table containing all relevant data
TABLE <- as.data.frame(cbind(quantiles,mean,sd,SQ,SUM,sem))
#####plotting results###
x11()
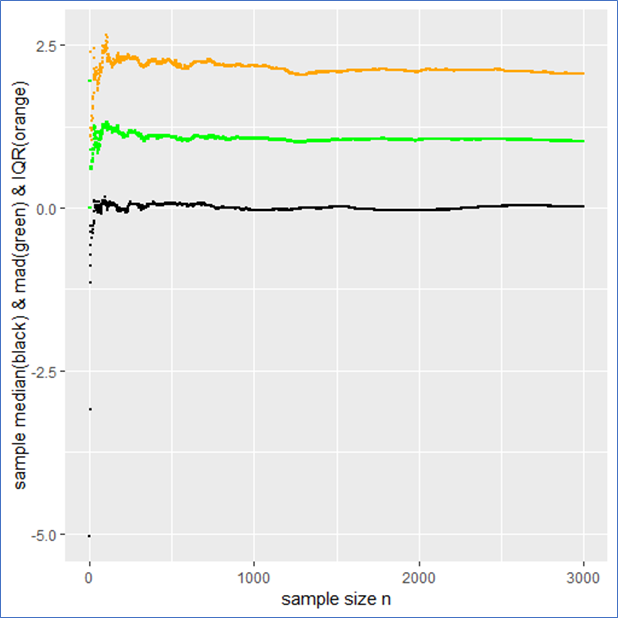
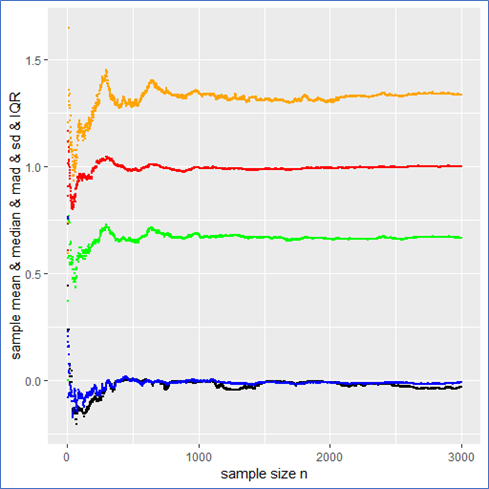
print(ggplot(TABLE,aes(1:N,median))+
geom_point(size=.5)+xlab('sample size n')+ylab('sample median'))
x11()
print(ggplot(TABLE,aes(1:N,mad))+geom_point(size=.5,color ='green')+
xlab('sample size n')+ylab('sample median absolute difference'))
x11()
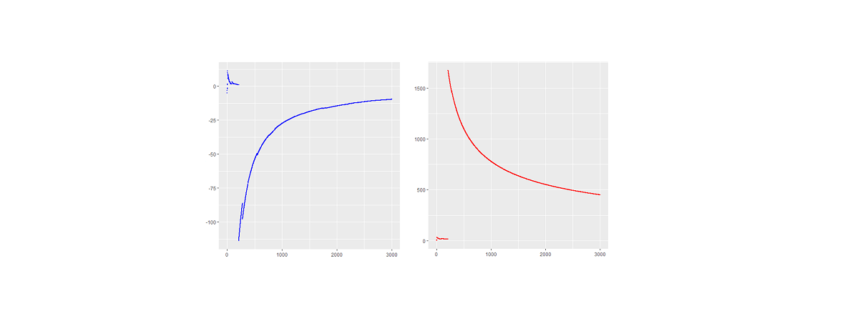
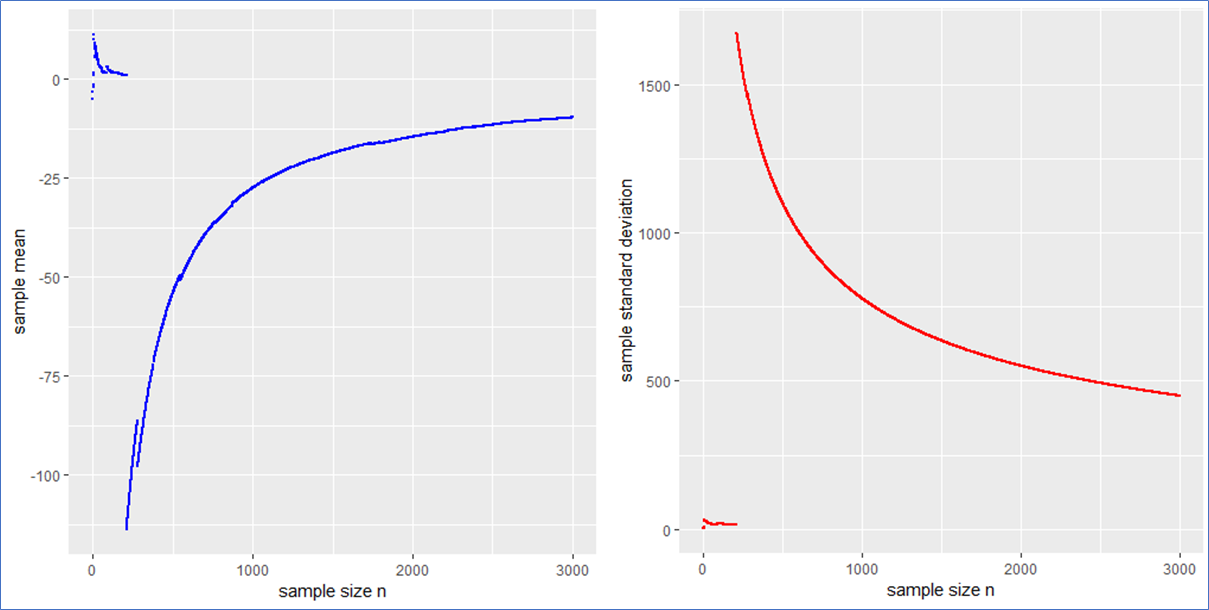
print(ggplot(TABLE,aes(1:N,sd))+geom_point(size=.5,color ='red')+
xlab('sample size n')+ylab('sample standard deviation'))
x11()
print(ggplot(TABLE,aes(1:N,mean))+geom_point(size=.5, color ='blue')+
xlab('sample size n')+ylab('sample mean'))
x11()
print(ggplot(TABLE,aes(1:N,Q3-Q1))+geom_point(size=.5, color ='blue')+
xlab('sample size n')+ylab('IQR'))
#uncomment the following lines of code to see further plots
#x11()
#print(ggplot(TABLE,aes(1:N,sem))+geom_point(size=.5)+
#xlab('sample size n')+ylab('sample sum of r.v.'))
#x11()
#print(ggplot(TABLE,aes(1:N,SUM))+geom_point(size=.5)+
#xlab('sample size n')+ylab('sample sum of r.v.'))
#x11()
#print(ggplot(TABLE,aes(1:N,SQ))+geom_point(size=.5)+
#xlab('sample size n')+ylab('sample sum of squares'))