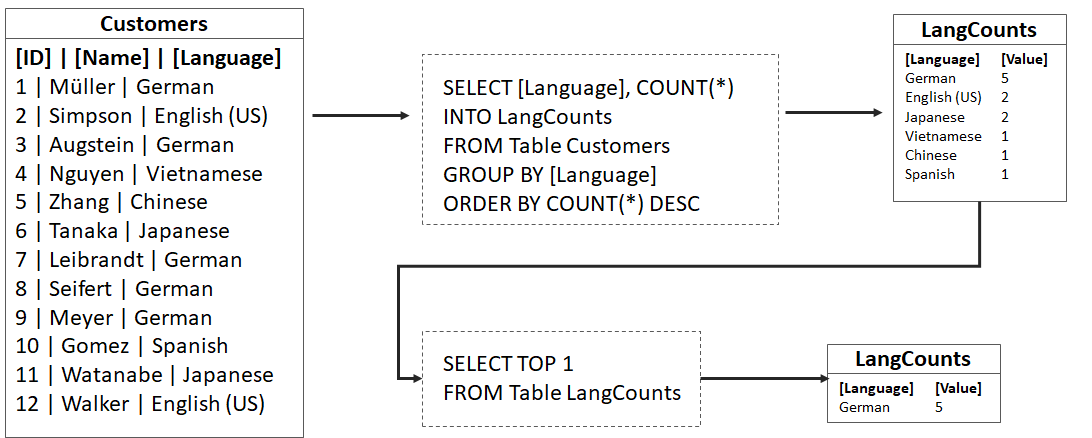
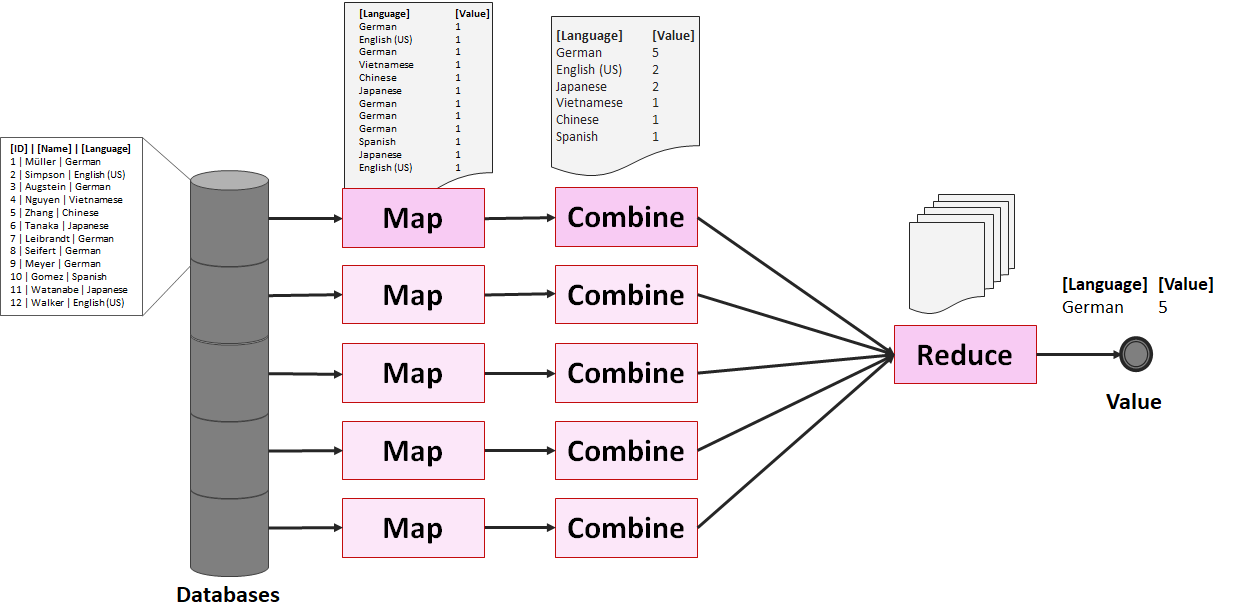
Wie kommen Daten die man via Internet quer durch die Welt sendet eigentlich an ihr Ziel? Welchen Weg nehmen beispielsweise die Datenpakete, wenn ich von mir zu Hause eine Datei an meinen Nachbarn ein Haus weiter sende? Wie groß ist der “Umweg”, den die Daten nehmen? Und macht es eigentlich einen Unterschied, ob ich www.google.de, www.google.com oder www.google.nl aufrufe, oder gehen alle Suchanfragen sowieso an dasselbe Ziel?
Fragen wie diese lassen sich durch eine Kombination von Tools wie traceroute oder tracepath und geoiplookup beantworten und unter Verwendung des Python-Paketes geoplotlib sogar graphisch auf einer Weltkarte darstellen. Die so gewonnenen Ergebnisse zeigen Teile der Netzwerktopologie des Internets auf und führen zu interessanten, teils unerwarteten Erkenntnissen.
Ziel dieses Artikels soll sein, ein möglichst einfaches Tutorial zum selber mitbasteln bereit zu stellen. Die einzelnen Schritte die hierfür notwendig sind, werden möglichst einfach verständlich dargestellt und erklärt, trotzdem sind zum vollständigen Verständnis grundlegende Kenntnisse in Python sowie der Kommandozeile hilfreich. Er richtet sich aber auch an alle, die sich einfach einmal etwas in ihrer virtuellen Umgebung „umschauen“ möchten oder einfach nur an den Ergebnissen interessiert sind, ohne sich mit den Details und wie diese umgesetzt werden, auseinander setzen zu wollen. Am Ende des Artikels werden die einzelnen Skripte des Projekts als zip-Datei bereitgestellt.
Hinweis: Diese Anleitung bezieht sich auf ein Linux-System und wurde unter Ubuntu getestet. Windows-User können beispielsweise mit dem Befehl tracert (als Ersatz für traceroute) ähnliche Ergebnisse erziehlen, jedoch muss dann das Parsing der IP-Adressen abgeändert werden.
1. Grundsätzliches Erkunden der Route, die ein Datenpaket nimmt
Hierfür wird ein Programm wie traceroute, tracepath oder nmap benötigt, welches durch Versenden von „abgelaufenen Datenpaketen“ die Hosts „auf dem Weg“ zum Ziel dazu bringt, ihre IPv4-Adresse zurück zu geben. In diesem Artikel wird beispielhaft traceroute verwendet, da dieses unter den meisten Linux-Versionen bereits zur „Grundausstattung“ gehört und somit für diesen Schritt keine weitere Software installiert werden muss. Die Verwendung von traceroute folgt der Syntax:
Als Ziel muss hier die IP-Adresse bzw. der Domainname des Zielrechners angegeben werden. Ein Beispiel soll dies vereinfachen:
|
|
$ sudo traceroute www.google.de traceroute to www.google.de (172.217.22.99), 64 hops max 1 192.168.0.1 167,148ms 3,200ms 11,636ms 2 83.169.183.11 21,389ms 19,380ms 88.134.203.107 16,746ms 3 88.134.203.107 27,431ms 24,063ms * 4 88.134.237.6 1679,865ms * 130,818ms 5 88.134.235.207 58,815ms 84,150ms * 6 72.14.198.218 144,998ms 107,364ms 108.170.253.68 121,851ms 7 108.170.253.84 58,323ms 101,127ms 216.239.57.218 44,461ms 8 216.239.57.218 43,722ms 91,544ms 172.253.50.100 67,971ms 9 172.253.50.214 106,689ms 96,100ms 216.239.56.130 110,334ms 10 209.85.241.145 63,720ms 61,387ms 209.85.252.76 73,724ms 11 209.85.252.28 71,214ms 61,828ms 108.170.251.129 81,470ms 12 108.170.251.129 64,262ms 52,056ms 72.14.234.115 71,661ms 13 72.14.234.113 262,988ms 55,005ms 172.217.22.99 66,043ms |
Im Beispiel wird die Route zum Hostrechner mit der Domain www.google.de ermittelt. In der ersten Spalte der Ausgabe ist die Nummer des jeweiligen „Hops“ zu sehen. Wichtig ist insbesondere die zweite Spalte, welche die IPv4-Adresse des jeweiligen Rechners auf dem Weg zum Ziel darstellt. Die folgenden Spalten enthalten weitere Informationen wie Antwortzeiten der jeweiligen Server und die IP-Adressen der Folge-Server.
Um die Ausgabe in eine Form umzuwandeln, welche später einfacher von Python gelesen werden kann, muss diese noch ausgelesen werden (Parsing). zuerst soll die erste Zeile der Ausgabe herausgeschnitten werden, da diese zwar informativ, jedoch kein Teil der eigentlichen Route ist. Dies kann sehr einfach durchgeführt werden, indem die Ausgabe des traceroute-Befehls an einen Befehl wie beispielsweise sed „gepiped“ (also weitergeleitet) wird. Die dabei entstehende Pipe sieht dann wie folgt aus:
|
|
sudo traceroute ${ZIEL} | sed '1d' |
Um bei unserem Beispiel mit der Route zu www.google.de zu bleiben, sieht der Befehl und die Entsprechende Ausgabe wie folgt aus:
|
|
$ sudo traceroute | sed '1d' 1 192.168.0.1 167,148ms 3,200ms 11,636ms 2 83.169.183.11 21,389ms 19,380ms 88.134.203.107 16,746ms 3 88.134.203.107 27,431ms 24,063ms * 4 88.134.237.6 1679,865ms * 130,818ms 5 88.134.235.207 58,815ms 84,150ms * 6 72.14.198.218 144,998ms 107,364ms 108.170.253.68 121,851ms 7 108.170.253.84 58,323ms 101,127ms 216.239.57.218 44,461ms 8 216.239.57.218 43,722ms 91,544ms 172.253.50.100 67,971ms 9 172.253.50.214 106,689ms 96,100ms 216.239.56.130 110,334ms 10 209.85.241.145 63,720ms 61,387ms 209.85.252.76 73,724ms 11 209.85.252.28 71,214ms 61,828ms 108.170.251.129 81,470ms 12 108.170.251.129 64,262ms 52,056ms 72.14.234.115 71,661ms 13 72.14.234.113 262,988ms 55,005ms 172.217.22.99 66,043ms |
Anschließend soll die zweite Spalte der Ausgabe herausgeschnitten werden. Dies ist am einfachsten mit dem Befehl awk zu bewerkstelligen. Das Prinzip dahinter ist das gleiche wie im obigen Schritt: die Ausgabe des vorherigen Befehls wird dem Befehl awk als Eingabe weitergeleitet, womit der gesamte Befehl nun wie folgt aussieht:
|
|
sudo traceroute ${ZIEL} | sed '1d' | awk '{ print $2 }' |
Bezogen auf das google-Beispiel sehen Ein- und Ausgabe nun so aus:
|
|
$ sudo traceroute | sed '1d' | awk '{ print $2 }' 192.168.0.1 83.169.183.11 88.134.203.107 88.134.237.6 88.134.235.207 72.14.198.218 108.170.253.84 216.239.57.218 172.253.50.214 209.85.241.145 209.85.252.28 108.170.251.129 72.14.234.113 |
Im letzten Schritt sollen die einzelnen IP-Adressen durch Leerzeichen getrennt in eine einzelne Zeile geschrieben werden. Sinn dieses Schrittes ist, dass später viele Zielrechner nacheinander aus einer Datei eingelesen werden können und jede Route zu einem Zielrechner als eine einzelne Zeile in eine Zieldatei geschrieben wird.
Auch dieser Schritt funktioniert ähnlich wie die obigen Schritte, indem die Ausgabe des letzten Schrittes an einen weiteren Befehl weitergeleitet wird, der diese Funktion erfüllt. Dieser Schritt könnte wieder mit dem Befehl sed durchgeführt werden, da aber nur ein einzelnes Zeichen (nämlich das Zeilenumbruch-Zeichen bzw. Newline) durch ein Leerzeichen ersetzt werden soll, wird hier aufgrund der einfacheren Syntax der Befehl tr verwendet.
Der fertige Befehl sieht nun wie folgt aus:
|
|
sudo traceroute ${ZIEL} | sed '1d' | awk '{ print $2 }' | tr '\n' ' ' |
Oder im fertigen Beispiel mit www.google.de:
|
|
$ sudo traceroute | sed '1d' | awk '{ print $2 }' | tr '\n' ' ' 192.168.0.1 83.169.183.11 88.134.203.107 88.134.237.6 88.134.235.207 72.14.198.218 108.170.253.84 216.239.57.218 172.253.50.214 209.85.241.145 209.85.252.28 108.170.251.129 72.14.234.113 |
Hiermit ist das Parsen abgeschlossen und die fertige Ausgabe kann nun in eine Ergebnisdatei geschrieben werden. Um automatisch viele Zielrechner aus einer Datei einzulesen und alle gefundenen Routen in eine Zieldatei zu schreiben, wird der obige Befehl in eine Schleife „verpackt“ welche die Zielrechner Zeile für Zeile aus der Datei zieladressen.txt ausliest und die gefundenen Routen ebenso Zeile für Zeile in die Datei routen.csv schreibt. Die Datei routen.csv kann später zur Ermittlung verschiedener Informationen zu den gefunden IP-Adressen einfach mit einem Python-Skript eingelesen und geparst werden.
In diesem Artikel wird das fertige Skript ohne weitere Erklärung in der beiliegenden zip-Datei bereitgestellt. Wen die genaue Funktionsweise der Schleife interessiert, sei angehalten sich generell über die Funktionsweise von Shellskripten einzulesen, da dies den Rahmen des Artikels sprengen würde.
|
|
#/bin/sh cat zieladressen.txt | while read ZIEL; do printf 'Ermittle Route nach: %s\n' "${ZIEL}" traceroute ${ZIEL} | sed '1d' | awk '{ print $2 }' | tr '\n' ' ' >> routes.csv printf '\n' >> routes.csv done cat routes.csv | tr -d \* | tr -s ' ' > routes_corrected.csv mv routes_corrected.csv routes.csv |
Dieses Skript benötigt die Datei zieladressen.txt welche wie folgt aussehen muss (anstatt Domainnamen können auch direkt IPv4-Adressen verwendet werden):
|
|
www.google.de www.github.com www.google.nl ... |
2. Sammeln von (Geo-)Informationen zu bestimmten IPv4-Adressen
Die gefundenen IPv4-Adressen können anschließend mit dem Befehl geoiplookup oder über die Internetseite http://geoiplookup.net/ relativ genau (meißtens auf Städteniveau) lokalisiert werden. Dies funktioniert, da einzelne Subnets in der Regel bestimmten Regionen und Internetprovidern zugeordnet sind.
Der Befehl geoiplookup greift hierbei auf eine vorher installierte und lokal gespeicherte Datenbank zu, welche je nach installierter Version als Country- oder City-Edition vorliegt. Da geoiplookup nicht zu den Standartbordmitteln unter Linux gehört und um die weiteren Schritte auch Benutzern anderer Betriebssysteme zu ermöglichen, wird hier nur ein kurzes Beispiel der Benutzung dieses Befehls und dessen Ausgabe gegeben und im weiteren die Online-Abfrage mittels eines Python-Skriptes beschrieben.
|
|
$ geoiplookup 172.217.22.99 GeoIP Country Edition: US, United States GeoIP City Edition, Rev 1: US, CA, California, Mountain View, 94043, 37.419201, -122.057404, 807, 650 GeoIP ASNum Edition: AS15169 Google Inc. |
Die Internetseite http://geoiplookup.net bietet einen Onlineservice welcher Geo- und weitere Informationen zu gegebenen IPv4-Adressen bereitstellt. Öffnet man die Seite ohne Angabe einer IP-Adresse in einem Browser, so erhält man die entsprechenden Informationen über die eigene IP-Adresse. (Achtung: die Verwendung eines Proxies oder gar Tor führt zwangsläufig zu falschen Ergebnissen.)
Da die Seite auch über eine API (also eine automatisierte Abfrageschnittstelle) unter der Adresse “http://api.geoiplookup.net/?query=${IPADRESSE}” verfügt, kann man die entsprechenden Informationen zu den IP-Adressen mittels eines Pythonskriptes abfragen und auswerten. Als Antwort erhält man eine XML‑Datei welche beispielsweise folgendermaßen aussieht:
|
|
77.20.253.87 77.20.253.87 Vodafone Kabel Deutschland Hamburg DE Germany 53.61530 10.1162 |
Diese kann im Browser z. B. unter der Adresse http://api.geoiplookup.net/?query=77.20.253.87 aufgerufen werden (oder unter: http://api.geoiplookup.net/ für die eigene Adresse).
Um die hierin enthaltenen Informationen mit Hilfe von Python auszulesen lässt sich ElementTree aus aus dem Modul xml.etree, das in der Python-Standartbibliothek vorhanden ist, verwenden. Dies wird im beiliegenden Skript mit der Funktion get_hostinfo() bewerkstelligt:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 |
def get_hostinfo(ipv4): ''' Returns geoiplookup information of agiven host adress as a dictionary. The adress can be given as a string representation 0f a DNS or IPv4 adress. get_hostinfo(str) -> dict Examples: get_hostinfo("www.github.com") get_hostinfo("151.101.12.133") ''' apiurl = 'http://api.geoiplookup.net/?query=' hostinfo = defaultdict(str, {}) try: xml = urllib.request.urlopen(apiurl + dns2ipv4(ipv4)).read().decode() xml = xml.replace('&', '') tree = ETree.fromstring(xml) for element in tree.getiterator(): hostinfo[element.tag] = element.text except: return hostinfo finally: return hostinfo |
Diese parst die XML-Datei automatisch zu einem Python-DefaultDict das dann die entsprechenden Informationen enthält (das DefaultDict wird verwendet da normale Python Dictionaries zu Fehlern führen, wenn nicht gesetzte Werte abgefragt werden). Die Ausgabe der Funktion sieht dann wie folgt aus:
|
|
In [3]: get_hostinfo('www.google.com') Out[3]: defaultdict(str, {'city': 'Mountain View', 'countrycode': 'US', 'countryname': 'United States', 'host': '172.217.22.99', 'ip': '172.217.22.99', 'isp': 'Google', 'latitude': '37.4192', 'longitude': '-122.0574', 'result': None, 'results': None}) |
3. Plotten der gefundenen Routen mit geoplotlib auf einer Weltkarte
Wichtig für das anschließende Plotten ist hierbei die Geolocation also ‘latitude’ und ‘longitude’. Mit den Werten kann man anschließend die mit traceroute gefundenen Pfade als Basemap plotten. Dies funktioniert mit der Funktion drawroutes2map():
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 |
def drawroutes2map(routesfile='routes.csv'): drawroutes = list() for route in open(routesfile).readlines(): ips = [ip2location(ip) for ip in route.strip().split(',')] print(ips) locs = [loc for loc in ips if not loc == None] longs = [loc[0] for loc in locs] lats = [loc[1] for loc in locs] m = minimalmap() drawroutes.append(tuple(m(lats, longs))) for drawroute in drawroutes: m.plot(drawroute[0], drawroute[1], '-', markersize=0, linewidth=1, color=rand_color()) pickleto(drawroutes, 'tracedlocs.plk') plt.savefig('world.svg', format='svg') plt.savefig('world.png', format='png') plt.show() |
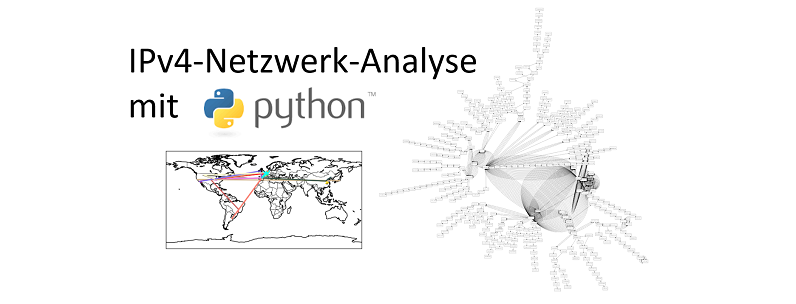
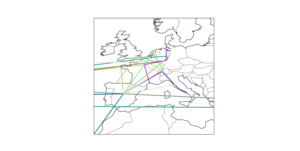
Der Plot einer Verbindungsanfrage an www.google.de aus Berlin sieht beispielsweise folgendermaßen aus:
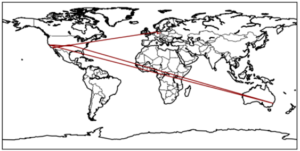
Hier wird deutlich, dass Datenpakete durchaus nicht immer den kürzesten Weg nehmen, sondern teilweise rund um die Welt gesendet werden (Deutschland – USA – Sydney(!) – USA), bevor sie an ihrem Ziel ankommen und dass das Ziel einer Verbindung zu einer Domain mit der Endung „de“ nicht unbedingt in Deutschland liegen muss.
Mit Default-Einstellungen werden von der Funktion drawroutes2map() alle Routen in zufälligen Farben geplottet, welche in der Datei routen.csv gefunden werden.
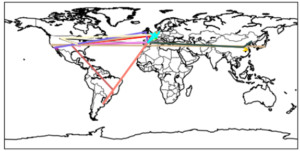
Lässt man viele Routen plotten wird hierbei die Netzwerkstruktur deutlich, über die die Daten im Internet verteilt werden. Auf dem obigen Plot kann man recht gut erkennen, dass die meisten Internetseiten in Europa oder den USA gehostet werden, einige noch in China und Japan, dagegen beispielsweise Afrika praktisch unbedeutend ist.
Auf dem nächsten Plot wiederum ist zu erkennen, dass es tatsächlich eine Art “Hotspots” gibt über die fast alle Daten laufen, wie z. B. Frankfurt am Main, Zürich und Madrid.
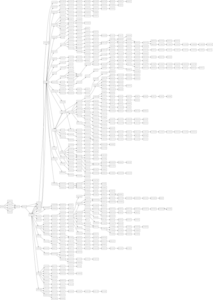
4. Schematische Darstellung der Routen als directed Graph mit graphviz
Mit graphviz lassen sich schematische Graphen darstellen. Mit dem Paket pygraphviz existiert hiefür auch eine Python-Anbindung. Die schematische Darstellung als Graph ist in vielen Fällen deutlich übersichtlicher als die Darstellung auf einer Weltkarte und die Topologie des Netzwerkes wird besser sichtbar.
Die entsprechende Python-Funktion, die alle Routen aus der Datei routes.csv als geplotteten Graph ausgibt ist drawroutes2graph():
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 |
def drawroutes2graph(routesfile='routes.csv'): '''Draws all routes found in the routesfile with graphviz to a Graph drawroutes2graph(file) ''' routes = open(routesfile).readlines() for i in range(len(routes)): routes[i] = routes[i].replace('*', '').split() G = pgv.AGraph(strict=False, directed=True) for l in routes: for i in range(len(l)-1): if not (l[i], l[i+1]) in set(G.edges()): G.add_edge(l[i], l[i+1]) for n in G.nodes(): if get_hostinfo(n)['countrycode'] == 'DE': n.attr['color'] = 'green' elif get_hostinfo(n)['countrycode'] == 'US': n.attr['color'] = 'red' elif get_hostinfo(n)['countrycode'] == 'ES': n.attr['color'] = 'yellow' elif get_hostinfo(n)['countrycode'] == 'CH': n.attr['color'] = 'blue' elif get_hostinfo(n)['countrycode'] == 'CN': n.attr['color'] = 'magenta' G.write('routes.dot') G.layout('dot') G.draw('dot.png') G.layout() G.draw('neato.png') |
Die Funktion schreibt den erstellten Graph in der Dot-Language in die Datei routes.dot und erstellt zwei verschiedene visuelle Darstellungen als png-Dateien.
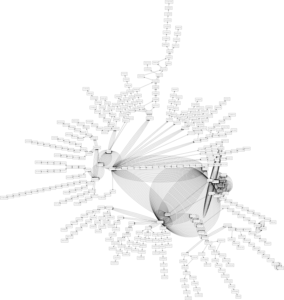
Da mit der Funktion get_hostinfo() auch weitere Informationen zu den jeweiligen IP-Adressen verfügbar sind können diese auch visuell im Graph dargestellt werden. So sind in der folgenden Darstellung Hosts in verschiedenen Ländern in unterschiedlichen Farben dargestellt. (Deutschland in grün, USA in rot, Spanien in gelb, Schweiz in blau, China in magenta und alle übrigen Länder und Hosts ohne Länderinformation in schwarz).
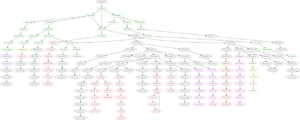
Diese Art der Darstellung vereint damit die Vorteile der schematischen Darstellung mit der Geoinformation zu den jeweiligen Hosts. Aus der Grafik lässt sich beispielsweise sehr gut erkennen, dass, trotz oft vieler Zwischenstationen innerhalb eines Landes, Landesgrenzen überschreitende Verbindungen relativ selten sind.
Auch interessant ist, dass das Netzwerk durchaus Maschen aufweist – mit anderen Worten: Dass ein und dieselbe Station bei verschiedenen Verbindungsanfragen über verschiedene Zwischenstationen angesprochen wird und Daten, die von Punkt A nach Punkt B gesendet werden, nicht immer denselben Weg nehmen.
5. Schlussfolgerung
Was kann man hieraus denn nun letztendlich an Erkenntnissen ziehen? Zum einen natürlich, wie Daten via Internet über viele Zwischenstationen rund um die Welt gesendet und hierbei mit jeder Station neu sortiert werden. Vor allem aber auch, dass mit dem entsprechenden Know-How und etwas Kreativität mit bemerkenswert wenig Code bereits Unmengen an Daten gesammelt, geordnet und ausgewertet werden können. Alle möglichen Daten werden in unserer heutigen Welt gespeichert und sind zu einem nicht unbeträchtlichen Teil auch für jeden, der weiß, wer diese Daten hat oder wie man sie selber ermitteln kann, verfügbar und oft lassen sich hier interessante Einblicke in die Funktionsweise unserer Welt gewinnen.