Distributed Computing – MapReduce Algorithmus
Sollen große Datenmengen analysiert werden, ist die Hardware eines leistungsfähigen Computers schnell überfordert und die Analysezeiten werden zu lang. Die Lösung zur Bewältigung von Big Data Analytics sind Konzepte des verteilten Rechnens (Distributed Computing).
Vertikale Skalierung – Der Klassiker der leistungsstarken Datenverarbeitung
Die meisten Unternehmen setzen heute noch auf leistungsstarke und aufrüstbare Einzelserver. Sollten Datenmengen größer und Analysen rechenaufwändiger werden, werden Festplatten (Storage), Arbeitsspeicher (RAM) und Prozessoren (CPU) aufgerüstet oder der Server direkt durch einen leistungsstärkeren ersetzt.
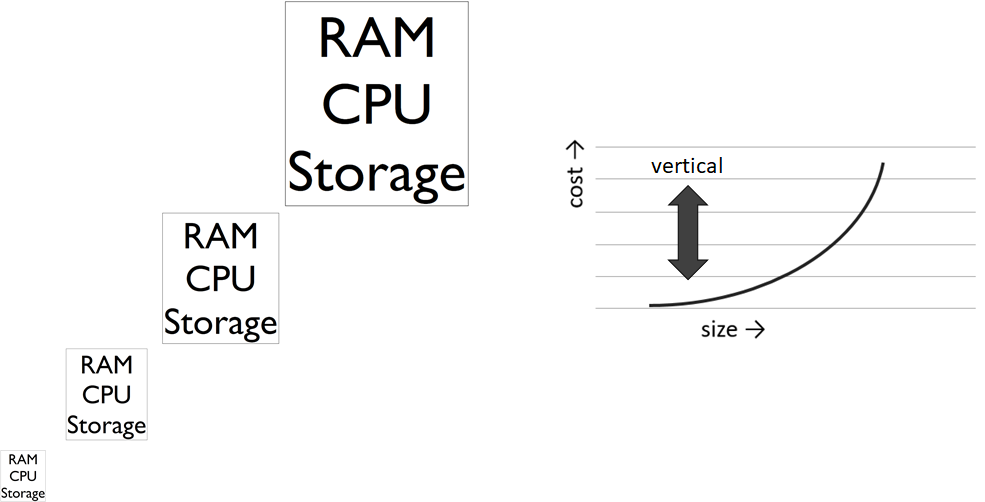
Diese Form der sogenannten vertikalen Skalierung (Vergrößerung der Server-Komponenten) ist für viele Unternehmen heute noch gängige Praxis, auch weil sie leicht zu administrieren ist und sie mit nahezu jeder Software funktioniert. Jedoch sind der Erweiterbarkeit gewisse Grenzen gesetzt und auch der Wechsel zu noch leistungsfähigerer Hardware würde den Einsatz von neuester High-End-Hardware bedeuten, der Kostenanstieg wäre exponentiell. Ferner bedarf es einer durchdachten Backup-Strategie mit gespiegelten Festplatten oder einem ganzen Backup-Server.
Leistungsstarke Server sind teuer und können zwar große Datenmengen weitaus schneller auswerten als Consumer-Computer, jedoch sind auch sie eher nicht dazu in der Lage, Big Data zu verarbeiten, also beispielsweise 100 Terabyte Daten binnen Sekunden statistisch auszuwerten.
Horizontale Skalierung – Skalierbare Speicher-/Rechenleistung
Ein alternatives Konzept zur vertikalen Skalierung ist die horizontale Skalierung. Dabei werden mehrere Computer, die im Vergleich oftmals über nur mittelmäßige Leistungsmerkmale verfügen, über ein Computer-Netzwerk verbunden und parallel angesteuert.
![]()
Der große Vorteil der horizontalen Skalierung ist der kostengünstige Einstieg, denn praktisch könnte bereits mit einem einzelnen Computer (Node) begonnen werden und dann nach und nach mit weiteren Nodes die Leistungsfähigkeit des Clusters (Verbund von Nodes) linear gesteigert werden. Ungefähr linear wachsen auch die Kosten an, so dass diese weitaus besser planbar sind. Cluster können weitaus höhere Leistungen erreichen als es einzelne Server könnten, daher gibt die horizontale Skalierung als diejenige, die sich für Big Data Analytics eignet, denn sie ermöglicht verteiltes Rechnen (Distributed Computing). Mit einem ausreichend großen Cluster lassen sich auch 100 Terabyte und mehr in wenigen Augenblicken statistisch auswerten.
Ferner ermöglichen horizontale Lösungen integrierte Backup-Strategien, indem jeder Node des Clusters über ein Backup der Daten eines anderen Nodes verfügt. Verfügt ein Node sogar über mehrere Backups, lässt sich eine sehr hohe Ausfallsicherheit – Datenverfügbarkeit im Cluster – erzielen.
Jedoch gibt es auch Nachteile der horizontalen Skalierung: Die Administration eines Clusters ist weitaus herausfordernder als ein einzelner Server, egal wie leistungsstark dieser sein mag. Auch Bedarf es viel räumlichen Platz für einen (oder gar mehrere) Cluster. Die Kompatibilität der Nodes sollte auch für die nächsten Jahr gesichert sein und nicht zuletzt ist es eine große Hürde, dass die einzusetzende Software (Datenbank- und Analyse-Software) für den Einsatz auf Clustern geeignet sein muss. Verbreite Software-Lösungen für verteiltes Speichern und Rechnen kommen beispielsweise von der Apache Foundation als Open Source Software: Hadoop, Spark und Flink.
Map Reduce Processing
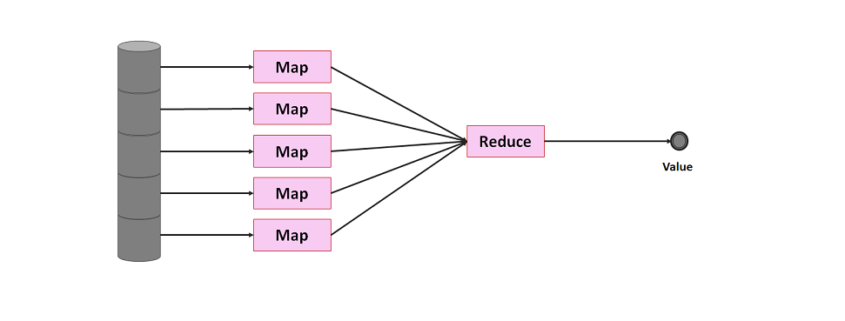
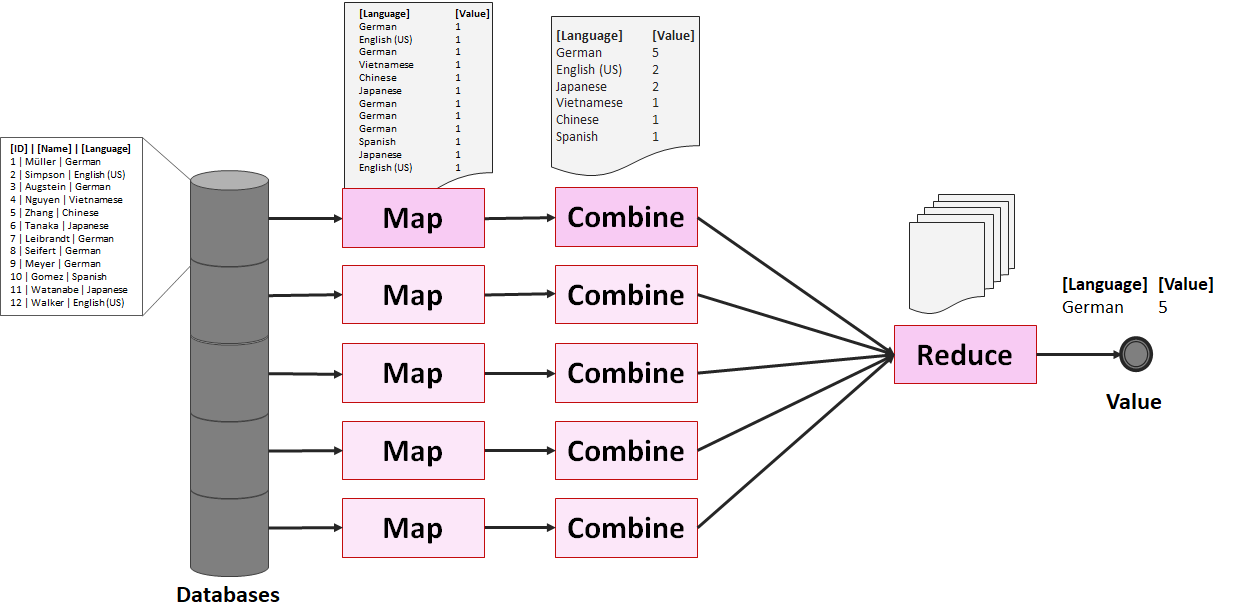
Damit verteiltes Rechnung funktioniert, bedarf es der richtigen Software, die wiederum Algorithmen einsetzt, die sich dafür eignen. Der bekannteste und immer noch am weitesten verbreitete Algorithmus ist MapReduce. MapReduce ist ein sehr einfacher Algorithmus und dürfte von der grundsätzlichen Vorgehensweise jedem Software-Entwickler oder Analyst vertraut sein. Das Prinzip entspricht dem folgenden SQL-Statement, dass die am häufigsten vorkommende Sprache aus dem Datensatz (Tabelle Customers) abfragt:
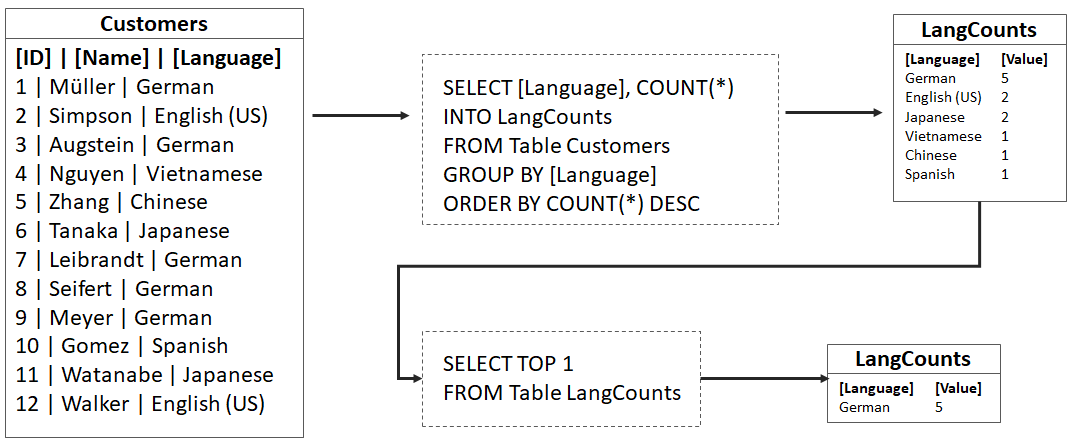
Es gibt eine Tabelle (es könnte eine Tabelle in einer relationalen Datenbank sein oder eine CSV-Datei), die durch eine SELECT-Query abgefragt (map), groupiert (combine) und sortiert (sort). Dieser Schritt kann vereinfacht als Map-Funktion betrachtet werden, die in einer Liste Paaren aus Schlüssel (Keys) und Werten (Values) resultiert. Ist diese Liste vorhanden, kann diese auf die gewünschten Ergebnisse entspechend einer Logik (z. B. max(), min(), mean(), sum()) auf wenige oder nur einen einzigen Wert reduziert werden (Reduce-Funktion). Zu beachten ist dabei, dass der Map-Prozess sehr viel speicher- und rechen-aufwändiger als der Reduce-Prozess ist. Führen wir diese Abfrage auf einer Maschine aus, fassen wir die beiden Abfragen als ein Statement aus:
SELECT TOP 1 [Language], COUNT(*) FROM Customers GROUP BY [Language] ORDER BY COUNT(*) DESC
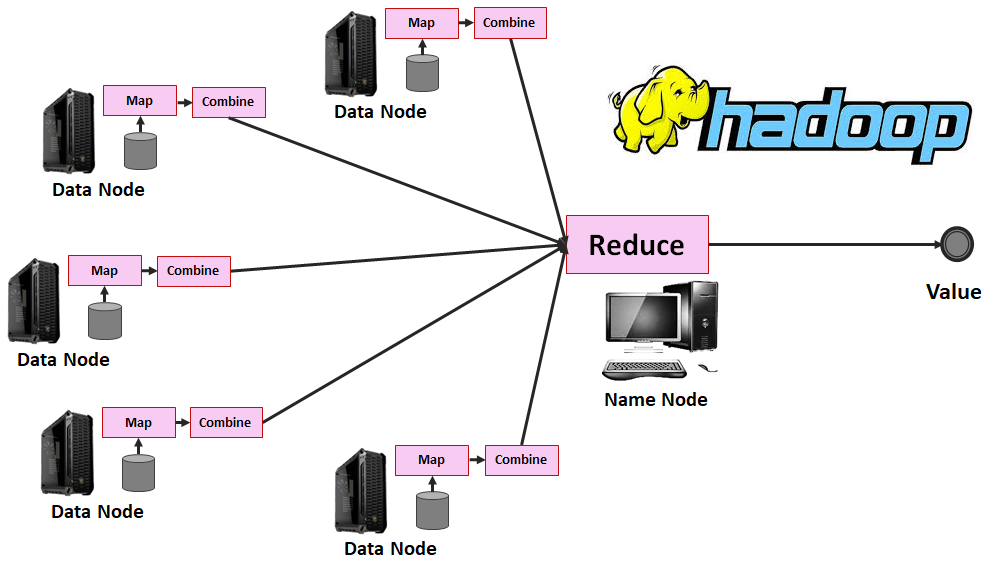
Betrachten wir jedoch die einzelnen Schritte, können wir sie wieder zumindest in einen Map- und einen Reduce-Schritt unterteilen. Diese Aufteilung machen wir uns für das verteilte Rechnen zunutze: Wenn jeder Computer (Node; oft auch Client Node oder Data Node) einen Teil der Daten besitzt, kann jeder Node für sich einen Map-Prozess durchführen, die Ergebnisse dann an einen Master-Node (oder in Hadoop-Sprache: Name Node) senden, der den Reduce-Prozess durchführt. Der Großteil der Aufgabe findet somit auf dem Cluster statt, nur der simple Reduce-Schritt auf einem einzelnen Computer.
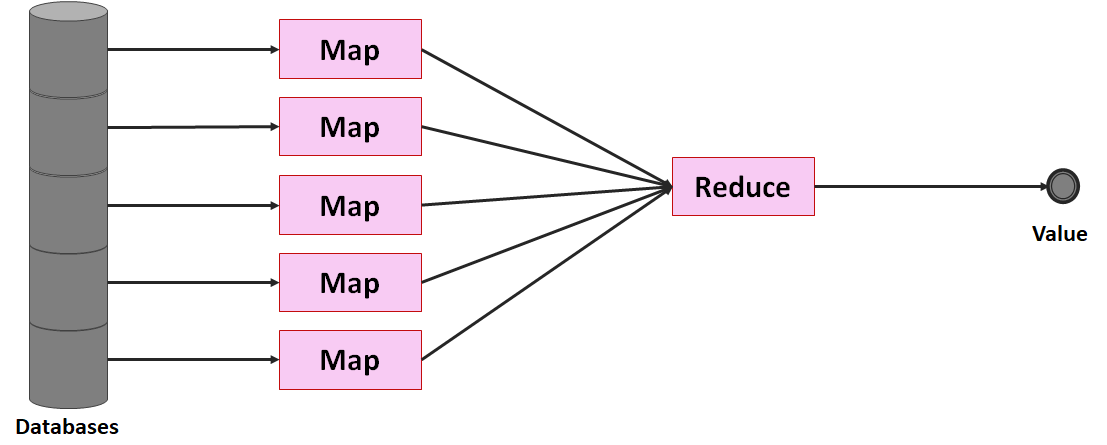
Oftmals reicht ein parallel ablaufender Map-Prozess auf dem Cluster nicht aus, um Daten effizient auswerten zu können. Die Maßgabe sollte stets sein, den Reduce-Aufwand so gering wie möglich zu halten und soviel Arbeit wie möglich auf den Cluster zu verlagern. Daher sollte jeder Node im Cluster soweit aggregieren wie möglich: Dafür gibt es den Combine-Schritt.
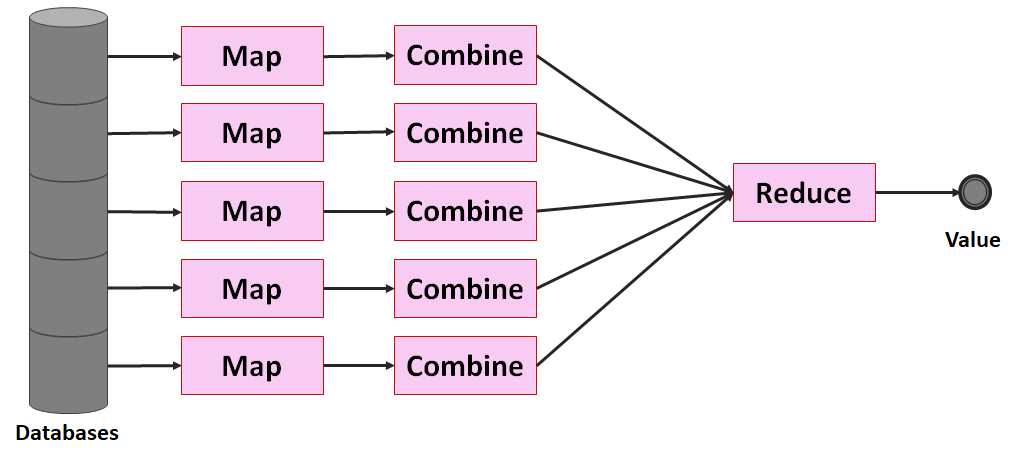
Die zuvor erwähnte SQL-Abfrage als MapReduce würde bedeuten, dass ein Node über den Datensatz verfügt (und andere Nodes über weitere Datensätze) und jeder Node für sich seine Daten über einen Map-Prozess herausarbeitet, über einen Combine-Prozess aggregiert und die Aggregationsergebnisse an den Master-Node (Name Node) sendet. Hat der Master-Node alle Ergebnisse erhalten, berechnet dieser daraus das Endergebnis (Reduce).
Zusammenfassung: Map Reduce
MapReduce ist der bekannteste Algorithmus zur verteilten Verarbeitung von Daten und eignet sich für die Durchführung von komplexen Datenanalysen. Liegen Datensätze auf mehreren Computern (Client Nodes) vor, läuft der Algorithmus in der Regel in drei Schritten ab:
- Map – Selektierung der Datensätze auf den Computern im gewünschten Format und Durchführung einer Berechnung, beispielsweise der Bildung einer Summe. Dieser Schritt ist ermöglich das Prinzip Schema on Read, das aus Hadoop ein Tool zur Verarbeitung von unstrukturierten Daten macht.
- Combine – Durchführung einer Aggregation, die ebenfalls auf jeden Client Node durchgeführt wird, zur Zusammenfassung von Map-Ergebnissen.
- Reduce – Aggregation aller Ergebnisse auf dem zentralen Rechner (Name Node)
MapReduce ist dazu geeignet, unstrukturierte Daten zu verarbeiten, denn das Format der Daten wird über einen Map-Algorithmus bestimmt, der sehr flexibel programmiert werden kann. MapReduce ist kein eng definierter Algorithmus, sondern eine Hülle, die mit Inhalt befüllt werden muss. So müssen MapReduce-Algorithmen individuell über eine Programmiersprache wie Java, Scala oder Python programmiert werden.
Ein Beispiel eines in Java programmierten Word-Count-Algorithmus nach der MapReduce-Logik in Hadoop findet sich hier:
1. package org.myorg;
2.
3. import java.io.IOException;
4. import java.util.*;
5.
6. import org.apache.hadoop.fs.Path;
7. import org.apache.hadoop.conf.*;
8. import org.apache.hadoop.io.*;
9. import org.apache.hadoop.mapred.*;
10. import org.apache.hadoop.util.*;
11.
12. public class WordCount {
13.
14. public static class Map extends MapReduceBase implements Mapper<LongWritable, Text, Text, IntWritable> { // Map-Process on Cluster
15. private final static IntWritable one = new IntWritable(1);
16. private Text word = new Text();
17.
18. public void map(LongWritable key, Text value, OutputCollector<Text, IntWritable> output, Reporter reporter) throws IOException {
19. String line = value.toString();
20. StringTokenizer tokenizer = new StringTokenizer(line);
21. while (tokenizer.hasMoreTokens()) {
22. word.set(tokenizer.nextToken());
23. output.collect(word, one);
24. }
25. }
26. }
27.
28. public static class Reduce extends MapReduceBase implements Reducer<Text, IntWritable, Text, IntWritable> { // Reduce-Process on Name Node
29. public void reduce(Text key, Iterator<IntWritable> values, OutputCollector<Text, IntWritable> output, Reporter reporter) throws IOException {
30. int sum = 0;
31. while (values.hasNext()) {
32. sum += values.next().get();
33. }
34. output.collect(key, new IntWritable(sum));
35. }
36. }
37.
38. public static void main(String[] args) throws Exception { // Setting up the MapReduce-Job "wordcount"
39. JobConf conf = new JobConf(WordCount.class);
40. conf.setJobName("wordcount");
41.
42. conf.setOutputKeyClass(Text.class);
43. conf.setOutputValueClass(IntWritable.class);
44.
45. conf.setMapperClass(Map.class);
46. conf.setCombinerClass(Reduce.class);
47. conf.setReducerClass(Reduce.class);
48.
49. conf.setInputFormat(TextInputFormat.class);
50. conf.setOutputFormat(TextOutputFormat.class);
51.
52. FileInputFormat.setInputPaths(conf, new Path(args[0]));
53. FileOutputFormat.setOutputPath(conf, new Path(args[1]));
54.
55. JobClient.runJob(conf);
57. }
58. }
MapReduce und Advanced Analytics
MapReduce spielt seine Vorteile auf Computer-Clustern aus und eignet sich sehr zur Analyse von Daten nach dem Schema on Read. Für kompliziertere Analysealgorithmen ist MapReduce jedoch nur bedingt geeignet, denn bereits einfache Join-Anweisungen benötigen mehrere MapReduce-Ketten.
Während statistische Auswertungen und Join-Anweisungen mit MapReduce noch gut möglich sind, werden Algorithmen des maschinellen Lernens schwierig bis kaum möglich, da diese viele Iterationen, z. B. zur Anpassung von Gewichten, benötigen.