Wahrscheinlichkeitsverteilungen – Zentralen Grenzwertsatz verstehen mit Pyhton
Wahrscheinlichkeitsverteilung sind im Data Science ein wichtiges Handwerkszeug. Während in der Mathevorlesung die Dynamik dieser Verteilungen nur durch wildes Tafelgekritzel schwierig erlebbar zu machen ist, können wir mit Programmierkenntnissen (in diesem Fall wieder mit Python) eine kleine Testumgebung für solche Verteilungen erstellen, um ein Gefühl dafür zu entwickeln, wie unterschiedlich diese auf verschiedene Wahrscheinlichkeitswerte, Varianz und Mengen an Datenpunkten reagieren und wann sie untereinander annäherungsweise ersetzbar sind – der zentrale Grenzwertsatz. Den Schwerpunkt lege ich in diesem Artikel auf die Binominal- und Normalverteilung.
Für die folgenden Beispiele werden folgende Python-Bibliotheken benötigt:
import matplotlib.pyplot as pyplot import random as random import math as math
Binominal-Verteilung
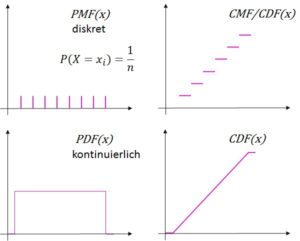
Die Binominalverteilung beschreibt die Wahrscheinlichkeiten von Bernoulli-Experimenten in ![]() endlichen Durchgängen. Bernoulli-Experimente sind solche, die eine binäre Ergebnissmenge haben (1 oder 0, Erfolg oder Misserfolg, …). Sie ist eine Wahrscheinlichkeitsmassefunktion (PMF – Probability Mass Function), die man grafisch an ihren Stufen (typisches Histogramm) erkennt.
endlichen Durchgängen. Bernoulli-Experimente sind solche, die eine binäre Ergebnissmenge haben (1 oder 0, Erfolg oder Misserfolg, …). Sie ist eine Wahrscheinlichkeitsmassefunktion (PMF – Probability Mass Function), die man grafisch an ihren Stufen (typisches Histogramm) erkennt.
Die Binominal-Verteilung gilt nur, wenn die Wahrscheinlichkeiten nach jedem Versuch gleich bleiben (also ein “Ziehen mit Zurücklegen”), andernfalls wäre die Hypergeometrische Verteilung anzuwenden.
Wenn man die Wahrscheinlichkeitsverteilungen richtig verstehen möchte, sollte man sich vor Augen führen, dass die Verteilungen in der Praxis selten genau so aussehen, wie es das mathematische Idealmodell vorgibt – insbesondere bei kleineren Datenmengen. Wir starten nun rein in die Programmierung mit einer kleinen Übung, über die wir eine (annähernde) Binominalverteilung unter Einsatz von Zufallszahlen künstlich erzeugen wollen.
Im Kern steht dabei das Bernoulli-Experiment, das ein binäres Ergebnis hat:
# Hier genieren wir einen Münz-/Würfelwurf (sogenannter Bernoulli-Versuch)
# ist der p-Wert hoch (z. B. 0.95), wrden die allermeisten Zufallszahlen
# kleiner oder gleich groß wie der p-Wert sein (etwa 95%).
# Umgekehrt, wenn der p-Wert klein ist, liegen entsprechend weniger Zufallszahlen
# im Bereich kleiner oder gleich des p-Wertes
def bernoulli_trial(p):
x = 1 if random.random() < p else 0
#print(x)
return x
Dieses Bernoulli-Exerperiment hat mit einer p-Wahrscheinlichkeit das Ergebnis 1, sonst 0. Die Ergebnismenge 0 oder 1 nutzen wir, um uns Zahlenmengen zu generieren. Wenn wir das Experiment x mal wiederholen, wie oft erhalten wir eine 1? Dies hängt von der Wahrscheinlichkeit ab, bei jedem Mal eine 1 zu erhalten. Dennoch kann sich die Menge natürlich jedes Mal trotz gleicher Wahrscheinlichkeit ändern!
def binomial(n, p):
bernoulli_trial_sum = sum(bernoulli_trial(p) for _ in range(n))
#print(bernoulli_trial_sum)
return bernoulli_trial_sum
Nachfolgend wiederhohen wir diese Experimente so viele Male, wie es uns der Parameter “num_points” vorgibt. Daraus erstellen wir uns ein Histogramm manuell, indem die gleichen Ergebnisse gezählt werden, beispielsweise könnte eine 23 (= 23 mal eine 1 “gewürfelt”) in der Menge acht mal vorkommen:
# Erstellt das Histogramm der Binominal-Verteilung
# als Wahrscheinlichkeitsmassefunktion aus den Zufallsversuchen heraus
def generateBinominalDistributionPMF(p, n, num_points):
data = [binomial(n, p) for _ in range(0, num_points)]
histogram = {}
for i in range(len(data)):
if data[i] in histogram:
histogram[data[i]] += 1;
else:
histogram[data[i]] = 1;
return histogram
Dieses Histogramm, das nichts weiter als ein Python-Dictionary ist, muss nun noch geplottet werden. Da wir uns das Histogramm manuell erstellt haben, nutzen wir nicht die .hist()-Funktion, sondern, da wir die y-Werte (= Menge des x-Ergebnisses) bereits haben, die .bar()-Funktion der matplotlib (Balkendiagramm).
def showHist(p, n, num_points):
# 3 x 3 Chart-Fenster für die Diagramme
# die Achsen-Objekte ax1, ax2 .. ax9 können zur Visualisierung verwendet werden
fig, ((ax1, ax2, ax3), (ax4, ax5, ax6), (ax7, ax8, ax9)) = pyplot.subplots(nrows=3, ncols=3, figsize=(15,10))
histogram = generateBinominalDistributionPMF(p, n, num_points)
ax1.bar([x for x in histogram.keys()], [v / num_points for v in histogram.values()], width = 1, color='red')
pyplot.show()
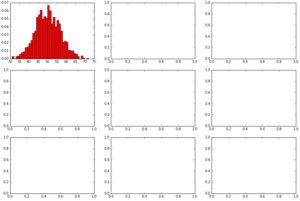
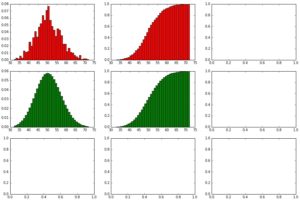
showHist(0.05, 1000, 1000)
Wir sehen die Binominal-Verteilung als erstes Chart, generiert aus den Bernoulli-Experimenten. Diese Verteilung sieht nach jedem Aufruf der showHist(0.05, 1000, 1000) jeweils etwas anders (einzigartig) aus und nicht wie eine perfekte Binominalverteilung nach dem mathematischen Modell, die wir uns weiter unten dann noch zum Vergleich dazu erstellen.
 Neben der Wahrscheinlichkeitsmasseverteilung (PMF) wird manchmal auch eine kumulierte Darstellung (CDF – Cumulative Distribution Function) benötigt, deren Kurve als Verteilungsfunktion beschrieben wird. Hierfür summieren wir die Zahlen aus der Datenreihe von links nach rechts auf.
Neben der Wahrscheinlichkeitsmasseverteilung (PMF) wird manchmal auch eine kumulierte Darstellung (CDF – Cumulative Distribution Function) benötigt, deren Kurve als Verteilungsfunktion beschrieben wird. Hierfür summieren wir die Zahlen aus der Datenreihe von links nach rechts auf.
# Erstellt das kumulierte Histogramm der Binominal-Verteilung
# basierend auf der Wahrscheinlichkeitsmassenfunktion
def generateBinominalDistributionCMF_by_PMF(histogram):
minKey = min(histogram.keys())
keylist = list(sorted(histogram.keys()))
histogramC = {}
for i in range(len(keylist)):
if keylist[i] == minKey:
histogramC[keylist[i]] = histogram[keylist[i]]
else: #if keylist[i] in histogram and keylist[i-1] in histogramC:
histogramC[keylist[i]] = histogram[keylist[i]] + histogramC[keylist[i-1]]
#print(i, keylist[i], histogram[keylist[i]], histogramC[keylist[i-1]], histogramC[keylist[i]])
return histogramC
Wir erweitern die showHist() entsprechend, so dass die Funktion aufgerufen und geplottet wird:
def showHist(p, n, num_points):
# 3 x 3 Chart-Fenster für die Diagramme
# die Achsen-Objekte ax1, ax2 .. ax9 können zur Visualisierung verwendet werden
fig, ((ax1, ax2, ax3), (ax4, ax5, ax6), (ax7, ax8, ax9)) = pyplot.subplots(nrows=3, ncols=3, figsize=(15,10))
histogram = generateBinominalDistributionPMF(p, n, num_points)
ax1.bar([x for x in histogram.keys()], [v / num_points for v in histogram.values()], width = 1, color='red')
histogramC = generateBinominalDistributionCMF_by_PMF(histogram)
ax2.bar([x for x in histogramC.keys()], [v / num_points for v in histogramC.values()], width = 1, color='red')
pyplot.show()
Nochmaliger Aufruf:
showHist(0.05, 1000, 1000)
Die kumulative Darstellung über das Balkendiagramm hat an dieser Stelle den Nachteil, dass manchmal in der Datenreihe (zwischen min(xs) und max(xs)) manchmal Werte gar nicht auftauchen (es wurde beispielsweise eine 45, 46 und 48 “gewürfelt”, aber keine 47), so dass hässliche Lücken im Diagramm entstehen, die aufzufüllen wären, würde es nun um die Ästhetik gehen 🙂
Modell der Binominal-Verteilung
Zuvor haben wir uns die Binominal-Verteilung als Wahrscheinlichkeitsmassefunktion (PMF) und als kumulierte Wahrscheinlichkeitsmassefunktion (CMF) aus Zufallszahlen heraus generiert. Die Wahrscheinlichkeit wird jedoch auch mathematisch als Modell beschrieben. Das ist nützlich, wenn man z. B. nur eine Stichprobe hat und eine Art “Schablone” benötigt, mit der es möglich ist, den Rest (Datenpunkte, die nicht in der Stichprobe enthalten sind) zu schätzen, ausgehend davon, dass der Rest der Logik der Wahrscheinlichkeitsverteilung folgt.
![]()
![]()
Der Erwartungswert für die Häufigkeit eines bestimmten Ergebnisses entspricht ![]() mit der Varianz
mit der Varianz ![]()
Wenn wir die Formel in den Pythoncode einhacken, sieht das z. B. so aus:
# Formel der Binominal-Verteilung (Wahrscheinlichkeitsmassefunktion)
def binominalDistributionPMF(k, n, p):
return (math.factorial(n) / (math.factorial(k) * math.factorial(n-k))) * p ** k * (1 - p)**(n-k)
Angenommen, Sie würfeln 10 mal einen Würfel (Ergebnissmenge: 1, 2, 3, 4, 5, 6), wie wahrscheinlich ist es, dass Sie 5 mal eine 6 würfeln (also Wahrscheinlichkeit 1/6 = 0,17)? Die Binominalverteilung verrät es Ihnen, ausgehend von der klassischen Wahrscheinlichkeitsbetrachtung (also Wahrscheinlichkeit 1/6 = 0,17):
print(binominalDistributionPMF(5, 10, 0.17)) # Ergebnis: 0.014094043564225089
Die Wahrscheinlichkeit bei zehn Würfen genau fünf mal eine 6 zu würfel, beträgt etwa 1,4%.
Wir plotten nun diese Formel über die Datenreihe xs, die wir uns zuvor generiert hatten:
def showBinominalDistributionPMF(xs, n, p, axis=None): ys = [binominalDistributionPMF(x, n, p) for x in xs] if axis == None: pyplot.bar(xs, ys, width=1, color="green") else: axis.bar(xs, ys, width=1, color="green")
Das ganze nochmal als CDF (bzw. CMF, wie die kumulierten Massefunktionen ab und an bezeichnet werden):
# Formel der Binominal-Verteilung (als Verteilungsfunktion) def showBinominalDistributionCMF(xs, n, p, axis=None): ys = [binominalDistributionPMF(x, n, p) for x in xs] for index, item in enumerate(ys): if index != 0: ys[index] += ys[index-1] if axis == None: pyplot.bar(xs, ys, width=1, color="green") else: axis.bar(xs, ys, width=1, color="green")
Nun noch den Aufruf der Funktionen in die showHist() einpflegen:
def showHist(p, n, num_points):
# 3 x 3 Chart-Fenster für die Diagramme
# die Achsen-Objekte ax1, ax2 .. ax9 können zur Visualisierung verwendet werden
fig, ((ax1, ax2, ax3), (ax4, ax5, ax6), (ax7, ax8, ax9)) = pyplot.subplots(nrows=3, ncols=3, figsize=(15,10))
histogram = generateBinominalDistributionPMF(p, n, num_points)
ax1.bar([x for x in histogram.keys()], [v / num_points for v in histogram.values()], width = 1, color='red')
histogramC = generateBinominalDistributionCMF_by_PMF(histogram)
ax2.bar([x for x in histogramC.keys()], [v / num_points for v in histogramC.values()], width = 1, color='red')
# Erstellung der Datenreihe basierend auf den zuvor erstellten Zufallsversuchen
xs = range(min(histogram), max(histogram) + 1)
showBinominalDistributionPMF(xs, n, p, axis=ax4)
showBinominalDistributionCMF(xs, n, p, axis=ax5)
pyplot.show()
Aufruf der Funktion wie zuvor:
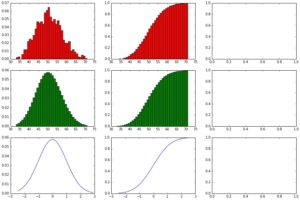
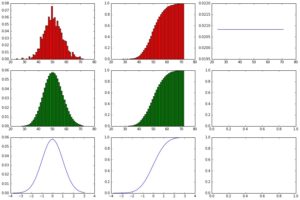
showHist(0.05, 1000, 1000)
Nun zeigt sich das perfekte mathematische Modell der Binominal-Verteilung im Vergleich zur zuvor via Zufallszahlen generierten Binominal-Verteilung.
Modell der Normalverteilung
Die Normalverteilung – ein absoluter Liebling der Statistiker – ist eine Wahrscheinlichkeitsverteilung, die in der Natur sehr häufig vorkommt. Beispielsweise ist die Körpergröße von Menschen in der Regel normalverteilt. Normalverteilt heißt grob erläutert, dass es sehr viele mittelgroße Menschen gibt, un nur ungefähr symmetrisch rechts und links davon immer weniger Ausprägungen gibt.
Die Normalverteilung zeigt sich nur bei sehr vielen Datenpunkten und ist entsprechend ihrer Stetigkeit eine Wahrscheinlichkeitsdichtefunktion (PDF – Probability Density Function), die im Gegensatz zur Wahrscheinlichkeitsmassefunktion keine sichtbaren Abstufungen hat, sondern als Linie dargestellt wird.
Die Formel der mathematischen Modellierung:
![]()
Umgesetzt im Python:
# Formel der Normalverteilung (als Wahrscheinlichkeitsdichtefunktion)
def normalDistributionPDF(x, mu=0, sigma = 1):
return (math.exp(-(x-mu) ** 2 / 2 / sigma ** 2) / ((math.sqrt(2 * math.pi) * sigma)))
Diese Funktion rufen wir nun einfach für alls x in der Datenreihe xs auf.
Optional ist die Darstellung als Standardnormalverteilung, die eine bessere Vergleichbarkeit ermöglicht. Dabei wird die erstellte Normalverteilung standardisiert, durch Umwandlng der x-Werte in z-Werte, in dem die Abweichung des x-Wertes zum Mittelwert ins Verhältnis zur Standardabweichung gesetzt wird:
![]()
Wir implementieren die Erstellung der Normalverteilung inklusive der optionalen Standardisierung:
def showNormalDistributionPDF(xs, mu=0, sigma=1, axis=None, standard=False, log=False): ys = [normalDistributionPDF(x, mu, sigma) for x in xs] # Standardisierung der Normalverteilung: if standard == True: xs = [(x - mu) / sigma for x in xs] if axis == None: pyplot.plot(xs, ys) else: axis.plot(xs,ys)
Auch die kumulative Darstellung (CDF – Cumulative Distribution Function) interessiert uns. Leider ist die mathematische Modellabbildung etwas hakelig. In der unten aufgeführten Literatur wird die Normalverteilung als CDF über die Nutzung der gaußschen Fehlerfunktion (erf() – error funktion) erstellt:
![]()
Wobei sich die Fehlerfunktion wie folgt beschreibt:
![]()
Die Fehlerfunktion soll uns gar nicht weiter stören, denn die math-Bibliothek in Python hält sie für uns parat:
# Formel der Normalverteilung (als Verteilungsfunktion) def normalDistributionCDF(x, mu = 0, sigma = 1): return (1 + math.erf((x - mu) / math.sqrt(2) / sigma)) / 2
Die Funktion zum Plotten wird ebenfalls implementiert …
def showNormalDistributionCDF(xs, mu = 0, sigma = 1, axis=None, standard=False): #ys = [normalDistributionCDF(i + 0.5, mu, sigma) - normalDistributionCDF(i - 0.5, mu, sigma) for i in xs] ys = [normalDistributionCDF(i, mu, sigma) for i in xs] if standard == True: xs = [(x - mu) / sigma for x in xs] if axis == None: pyplot.plot(xs, ys) else: axis.plot(xs,ys)
… und in die showHist() zum Aufruf eingetragen. Für den Aufruf brauchen wir noch den Mittelwert und die Standardabweichung der Normalverteilung für die Modelldarstellung, diese können wir über die Angaben n und p überschlagen:
def showHist(p, n, num_points):
# 3 x 3 Chart-Fenster für die Diagramme
# die Achsen-Objekte ax1, ax2 .. ax9 können zur Visualisierung verwendet werden
fig, ((ax1, ax2, ax3), (ax4, ax5, ax6), (ax7, ax8, ax9)) = pyplot.subplots(nrows=3, ncols=3, figsize=(15,10))
histogram = generateBinominalDistributionPMF(p, n, num_points)
ax1.bar([x for x in histogram.keys()], [v / num_points for v in histogram.values()], width = 1, color='red')
histogramC = generateBinominalDistributionCMF_by_PMF(histogram)
ax2.bar([x for x in histogramC.keys()], [v / num_points for v in histogramC.values()], width = 1, color='red')
# Erstellung der Datenreihe basierend auf den zuvor erstellten Zufallsversuchen
xs = range(min(histogram), max(histogram) + 1)
showBinominalDistributionPMF(xs, n, p, axis=ax4)
showBinominalDistributionCMF(xs, n, p, axis=ax5)
mu = p * n # Mittelwert
sigma = math.sqrt(n * p * (1 - p)) # Standardabweichung
showNormalDistributionPDF(xs, mu, sigma, axis=ax7, standard=True)
showNormalDistributionCDF(xs, mu, sigma, axis=ax8, standard=True)
pyplot.show()
Neuer Aufruf der showHist():
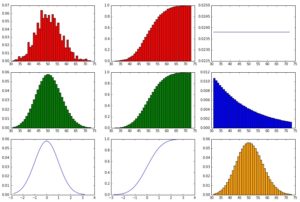
showHist(0.05, 1000, 1000)
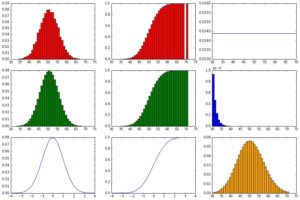
Nun sehen wir die Normalverteilung im Vergleich zur Binominalverteilung und erkennen schon den zentralen Grenzwertsatz, der im Kern die Aussage trifft, dass die Binominalverteilung bei größer werdenden Mengen an Datenpunkten sich der Normalverteilung annähert.
Modell der Gleichverteilung
Häufig in ihrer Bedeutung unterschätzt ist die Gleichverteilung, die mathematisch im Vergleich zur Normalverteilung quasi Entspannung pur ist. Wir können sie uns aus der Datenreihe künstlich erzeugen, indem wir ihren Wert einfach durch ihre Menge teilen. Die Gleichverteilung wird z. B. als Wahrscheinlichkeitsverteilung beim Hypothesentesting verwendet, um die Hypothese zu beweisen (oder zu widerlegen), dass ein Produkt in allen Regionen gleichhäufig verkauft wird.
![]()
Um das mathematische Modell der Gleichverteilung auf unsere Datenreihe anzuwenden, müssen wir nur für jedes ![]() nur
nur ![]() rechnen:
rechnen:
# Formel der Gleichverteilung def uniform(n): return 1/n
Wobei ![]() einfach die Anzahl an Datenpunkten ist:
einfach die Anzahl an Datenpunkten ist:
def uniformDistributionPDF(x): ys = [uniform(len(x)) for _ in x] return ys
Die Funktion zum Plotten:
def showUniformDistributionPDF(xs, axis=None): ys = uniformDistributionPDF(xs) if axis == None: pyplot.plot(xs, ys) else: axis.plot(xs, ys)
Eingepflegt in die showHist():
def showHist(p, n, num_points): # 3 x 3 Chart-Fenster für die Diagramme # die Achsen-Objekte ax1, ax2 .. ax9 können zur Visualisierung verwendet werden fig, ((ax1, ax2, ax3), (ax4, ax5, ax6), (ax7, ax8, ax9)) = pyplot.subplots(nrows=3, ncols=3, figsize=(15,10)) histogram = generateBinominalDistributionPMF(p, n, num_points) ax1.bar([x for x in histogram.keys()], [v / num_points for v in histogram.values()], width = 1, color='red') histogramC = generateBinominalDistributionCMF_by_PMF(histogram) ax2.bar([x for x in histogramC.keys()], [v / num_points for v in histogramC.values()], width = 1, color='red') # Erstellung der Datenreihe basierend auf den zuvor erstellten Zufallsversuchen xs = range(min(histogram), max(histogram) + 1) showBinominalDistributionPMF(xs, n, p, axis=ax4) showBinominalDistributionCMF(xs, n, p, axis=ax5) mu = p * n # Mittelwert sigma = math.sqrt(n * p * (1 - p)) # Standardabweichung showNormalDistributionPDF(xs, mu, sigma, axis=ax7, standard=True) showNormalDistributionCDF(xs, mu, sigma, axis=ax8, standard=True) showUniformDistributionPDF(xs, ax3) pyplot.show()
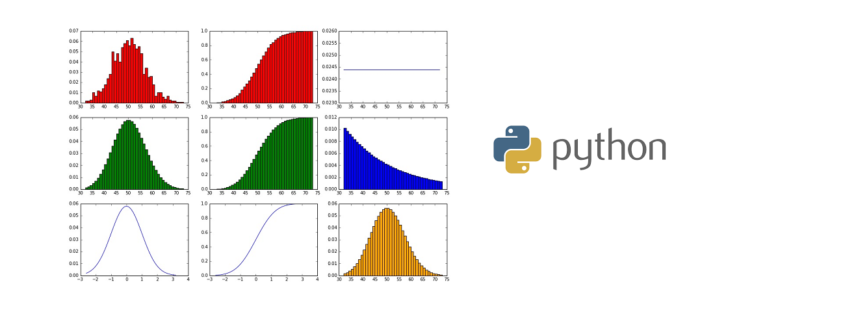
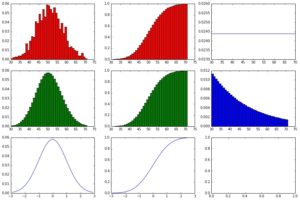
showHist(0.05, 1000, 1000)
Die Gleichverteilung ist als PDF (also als horizontale Gerade) oben rechts zu sehen.
Modell der Geometrischen Verteilung
Die Geometrische Verteilung ist verwandt mit der Binominal-Verteilung. Während die Binominal-Verteilung jedoch eine Wahrscheinlichkeitsmassefunktion zur Wahrscheinlichkeitsberechnung für das Auftreten eines Ereignisses in einer bestimmten Anzahl ist, kann mit der Geometrischen Verteilung berechnet werden, wie wahrscheinlich das erstmalige Auftreten des gesuchten Ereignisses nach ![]() -Versuchen ist.
-Versuchen ist.
![]()
Der Erwartungswert bedeutet, nach wie viel Versuchen wir mit dem ersten Erfolg “rechnen” können.
![]()
![]()
Umsetzung der Formel in Python:
# Formel der Geometrischen Verteilung (Wahrscheinlichkeitsmassefunktion) def geom(k, p): return p * (1 - p)**(k-1)
Aufruf der Funktion für alle x=k in xs:
def geomDistributionPMF(xs, p): ys = [geom(k, p) for k in xs] return ys
Plotten der Geometrischen Verteilung:
def showGeomDistributionPMF(xs, p, axis=None): ys = geomDistributionPMF(xs, p) if axis == None: pyplot.bar(xs, ys, width=1) else: axis.bar(xs, ys, width=1)
Einfügen des Funktionsaufrufes in die showHist():
def showHist(p, n, num_points): # 3 x 3 Chart-Fenster für die Diagramme # die Achsen-Objekte ax1, ax2 .. ax9 können zur Visualisierung verwendet werden fig, ((ax1, ax2, ax3), (ax4, ax5, ax6), (ax7, ax8, ax9)) = pyplot.subplots(nrows=3, ncols=3, figsize=(15,10)) histogram = generateBinominalDistributionPMF(p, n, num_points) ax1.bar([x for x in histogram.keys()], [v / num_points for v in histogram.values()], width = 1, color='red') histogramC = generateBinominalDistributionCMF_by_PMF(histogram) ax2.bar([x for x in histogramC.keys()], [v / num_points for v in histogramC.values()], width = 1, color='red') # Erstellung der Datenreihe basierend auf den zuvor erstellten Zufallsversuchen xs = range(min(histogram), max(histogram) + 1) showBinominalDistributionPMF(xs, n, p, axis=ax4) showBinominalDistributionCMF(xs, n, p, axis=ax5) mu = p * n # Mittelwert sigma = math.sqrt(n * p * (1 - p)) # Standardabweichung showNormalDistributionPDF(xs, mu, sigma, axis=ax7, standard=True) showNormalDistributionCDF(xs, mu, sigma, axis=ax8, standard=True) showUniformDistributionPDF(xs, ax3) showGeomDistributionPMF(xs, p, ax6) pyplot.show()
showHist(0.05, 1000, 1000)
Die Geometrische Verteilung ist in der mitte rechts in blau zu sehen. Da wir hier ein kleines p = 5% haben, ist die Erfolgswahrscheinlichkeit über die Versuche nicht steil fallend, die Varianz ist relativ klein.
Poisson-Verteilung
Die Poisson-Verteilung ist – auch wenn dieses grässliche Lambda (![]() ) den ersten Eindruck vermiest – von der Formel her recht einfach gestrickt.
) den ersten Eindruck vermiest – von der Formel her recht einfach gestrickt.
![]()
Der Erwartungswert ist einfach ![]() , was bedeutet, dass in
, was bedeutet, dass in ![]() bereits einiges Vorwissen stecken muss (nämlich ein durchschnittliches Auftreten eines Ereignisses in einer definierten stetigen Spanne). Mit der Poisson-Verteilung kann z. B. kalkuliert werden, wir wahrscheinlich ein Ausfall einer Maschine in der Produktion ist (allerdings nur theoretisch, die Weibull- und andere Wahrscheinlichkeitsfunktionen sind in der Praxis besser, da diese die Anlaufschwierigkeiten im Anfnagszeitraum und die Abnutzung nach dem Anlauf besser abbilden).
bereits einiges Vorwissen stecken muss (nämlich ein durchschnittliches Auftreten eines Ereignisses in einer definierten stetigen Spanne). Mit der Poisson-Verteilung kann z. B. kalkuliert werden, wir wahrscheinlich ein Ausfall einer Maschine in der Produktion ist (allerdings nur theoretisch, die Weibull- und andere Wahrscheinlichkeitsfunktionen sind in der Praxis besser, da diese die Anlaufschwierigkeiten im Anfnagszeitraum und die Abnutzung nach dem Anlauf besser abbilden).
Die Umsetzung der Formel in Python:
# Formel der Poisson-Verteilung def poisson(k, lambda_): return (math.exp(-1*lambda_) * lambda_**k) / math.factorial(k)
Wenn wir die Anzahl an Datenpunkten ![]() und die Wahrscheinlichkeit
und die Wahrscheinlichkeit ![]() kennen, können
kennen, können ![]() recht einfach ermitteln.
recht einfach ermitteln.
![]()
Dieses Wissen machen wir uns zu Nutze:
def showPoissonDistrubutionPMF(xs, n, p, axis=None): lambda_ = n*p ys = [poisson(x, lambda_) for x in xs] if axis == None: pyplot.bar(xs, ys, width=1, color="orange") else: axis.bar(xs, ys, width=1, color="orange")
Der Funktionsaufruf wird in die showHist() eingepflegt und diese wiederum mit den üblichen Parametern aufgerufen:
def showHist(p, n, num_points): # 3 x 3 Chart-Fenster für die Diagramme # die Achsen-Objekte ax1, ax2 .. ax9 können zur Visualisierung verwendet werden fig, ((ax1, ax2, ax3), (ax4, ax5, ax6), (ax7, ax8, ax9)) = pyplot.subplots(nrows=3, ncols=3, figsize=(15,10)) histogram = generateBinominalDistributionPMF(p, n, num_points) ax1.bar([x for x in histogram.keys()], [v / num_points for v in histogram.values()], width = 1, color='red') histogramC = generateBinominalDistributionCMF_by_PMF(histogram) ax2.bar([x for x in histogramC.keys()], [v / num_points for v in histogramC.values()], width = 1, color='red') # Erstellung der Datenreihe basierend auf den zuvor erstellten Zufallsversuchen xs = range(min(histogram), max(histogram) + 1) showBinominalDistributionPMF(xs, n, p, axis=ax4) showBinominalDistributionCMF(xs, n, p, axis=ax5) mu = p * n # Mittelwert sigma = math.sqrt(n * p * (1 - p)) # Standardabweichung showNormalDistributionPDF(xs, mu, sigma, axis=ax7, standard=True) showNormalDistributionCDF(xs, mu, sigma, axis=ax8, standard=True) showUniformDistributionPDF(xs, ax3) showGeomDistributionPMF(xs, p, ax6) showPoissonDistrubutionPMF(xs, n, p, axis=ax9) pyplot.show()
showHist(0.05, 1000, 1000)
Die Poisson-Verteilung ist im Chart unten rechts in gelb zu sehen. Auffällig ist die Ähnlichkeit zur Binominalverteilung und in der Tat wird die Poisson-Verteilung gerne dazu verwendet, um eine Binominal-Verteilung zu approximieren – Aber das funktioniert nur unter bestimmten Voraussetzungen (mehr dazu unten!).
Pareto-Verteilung
Die Pareto-Verteilung wurde vom Ökonomen Vilfredo Pareto beschrieben und ist die Grundlage für die Regel, dass 80% aller Werte von 20% aus der Population hervorgehen (und dass ich 80% dieses Artikels in 20% der Zeit erstellt habe, für die letzten 20% aber tatsächlich 80% der Zeit aufwende :-).
![]()
Die Paremeter ![]() (der kleinste Wert der Datenreihe) und
(der kleinste Wert der Datenreihe) und ![]() bestimmen die Lage und Form der Pareto-Verteilung.
bestimmen die Lage und Form der Pareto-Verteilung.
Die Pareto-Verteilung wird dazu verwendet, Einkommensverteilungen oder auch Größenverteilungen in der Natur zu beschreiben/schätzen/vorherzusagen. Wir erwarten eine Pareto-Verteilung beispielsweise bei der ABC-Analyse. Die meisten erkennen in der Verteilungsfunktion (CDF) direkt die typische Form der Verteilung der Materialwerte entsprechend der ABC-Analyse wieder.
![]()
Die Umsetzung in Python:
# Formel der Pareto-Verteilung def pareto(x, x_min, k, CDF): y = ((x/x_min)**(-k)) if CDF == True: y = 1 -y return y
Die Funktion zum Plotten der Pareto-Verteilung:
def showParetoDistributionPDF(xs, x_min, k, CDF=False, axis=None): ys = [pareto(x, x_min, k, CDF) for x in xs] if axis == None: pyplot.plot(xs, ys) else: axis.plot(xs,ys)
Der Aufruf als PDF in der showHist() (da ein zehntes Chart-Fenster fehlt, nun einfach im Austausch zur Poisson-Verteilung):
def showHist(p, n, num_points): # 3 x 3 Chart-Fenster für die Diagramme # die Achsen-Objekte ax1, ax2 .. ax9 können zur Visualisierung verwendet werden fig, ((ax1, ax2, ax3), (ax4, ax5, ax6), (ax7, ax8, ax9)) = pyplot.subplots(nrows=3, ncols=3, figsize=(15,10)) histogram = generateBinominalDistributionPMF(p, n, num_points) ax1.bar([x for x in histogram.keys()], [v / num_points for v in histogram.values()], width = 1, color='red') histogramC = generateBinominalDistributionCMF_by_PMF(histogram) ax2.bar([x for x in histogramC.keys()], [v / num_points for v in histogramC.values()], width = 1, color='red') # Erstellung der Datenreihe basierend auf den zuvor erstellten Zufallsversuchen xs = range(min(histogram), max(histogram) + 1) showBinominalDistributionPMF(xs, n, p, axis=ax4) showBinominalDistributionCMF(xs, n, p, axis=ax5) mu = p * n # Mittelwert sigma = math.sqrt(n * p * (1 - p)) # Standardabweichung showNormalDistributionPDF(xs, mu, sigma, axis=ax7, standard=True) showNormalDistributionCDF(xs, mu, sigma, axis=ax8, standard=True) showUniformDistributionPDF(xs, ax3) showGeomDistributionPMF(xs, p, ax6) #showPoissonDistrubutionPMF(xs, n, p, axis=ax9) showParetoDistributionPDF(range(1,100), 0.5, 0.5, CDF=False, axis=ax9) pyplot.show()
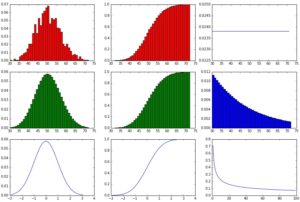
showHist(0.05, 1000, 1000)
Es zeigt sich die typische Pareto-Kurve im untersten rechten Diagramm-Bereich:
Verhalten der Wahrscheinlichkeiten bei Parameter-Änderung (p, n)
Nun schauen wir uns an, wie die Wahrscheinlichkeitsverteilungen auf unterschiedliche Größen p und n reagieren.
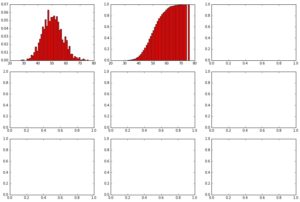
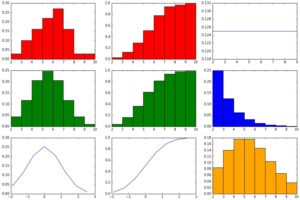
showHist(0.50, 10, 100)
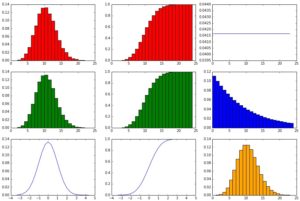
Wir haben hier eine Wahrscheinlichkeit für jeden Versuch 50% für das eine oder andere Ergebnis und nur eine kleine mögliche Ergebnismenge (0 – 10) sowie nur 100 Datenpunkte (also 100 mal einen Wert zwischen 0 und 10).
Die über Zufallszahlen generierte Binominal-Verteilung sieht (da per Zufall entstanden) nicht genau wie das Modell der Binominal-Verteilung aus, aber immerhin recht ähnlich. Die Normalverteilung nach Modell ist nicht richtig dargestellt (gaußsche Glocke ist nicht richtig ausgebildet), da die 100 Datenpunkte einfach zu wenige sind. Die Geometrische Verteilung nach Modell zeigt die 50/50-Wahrscheinlichkeit pro Versuch/Schritt sehr gut, denn die Balken halbieren sich genau jedes Mal. Die Poisson-Verteilung ähnelt der Binominal-Verteilung nur sehr entfernt bzw. gar nicht.
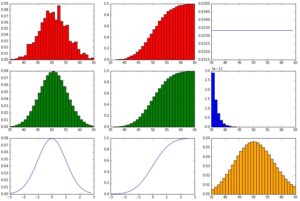
showHist(0.50, 100, 1000)
Immer noch eine 50%-Wahrscheinlichkeit, aber nun ein Ergebnisraum zwischen 0 und 100 (wobei genau 0 und 100 sehr unwahrscheinlich sind, da der Erwartungswert = 50). Und nun zehn mal mehr Datenpunkte als zuvor!
Die generierte Binominal-Verteilung ähnelt ihm Ideal-Modell immer noch nur ungefähr, die zufälligen Ausreißer werden vor allem in der PMF deutlich. Deutlicher wird nun die Ähnlichkeit zwischen dem Modell der Binominal-Verteilung und der Normalverteilung. Auch die Poisson-Verteilung scheint der Binominal-Verteilung ähnlicher zu werden, hat aber noch zu viel Varianz.
showHist(0.50, 100, 10000)
Immer noch 50%-Wahrscheinlichkeit und ein möglicher Ergebnisraum zwischen 0 und 100. Aber nochmal zehn mal mehr Datenpunkte.
Die generierte Binominal-Verteilung ähnelt ihm Ideal-Modell schon recht ordentlich, Ausreißer machen sich nur noch geringfügig erkennbar. Die Ähnlichkeit zwischen dem Modell der Binominal-Verteilung und der Normalverteilung ist nicht mehr von der Hand zu weisen.
showHist(0.10, 100, 10000)
Nun nur noch eine Wahrscheinlichkeit (für einen “Erfolg” bzw. eine 1 im Bernoulli-Experiment) von 10%. Der mögliche Ergebnisraum liegt zwar immer noch zwischen 0 und 100, aber es werden niedrigere Werte wahrscheinlicher.
Die Form der Binominal-Verteilung und der Normalverteilung hat sich nicht verändert, allerdings ist der Mittelwert stark nach links verschoben. Die Poisson-Verteilung scheint der Binominal-Verteilung sehr ähnlich geworden zu sein. Diese Erkenntnis ist wichtig, denn die Poisson-Verteilung wird manchmal dazu verwendet, die Binominal-Verteilung (die aufwändiger zu berechnen ist) zu approximieren. Dies funktioniert jedoch nur bei kleinen p-Werten und vielen Datenpunkten.
Auch die Geometrische Verteilung zeigt eine deutliche Reaktion, denn die Erfolgswahrscheinlichkeit liegt pro Wurf niedriger, der Abfall ist daher auch flacher (es ist nun unwahrscheinlicher, bereits nach dem ersten Durchgang gleich einen Erfolg zu haben).
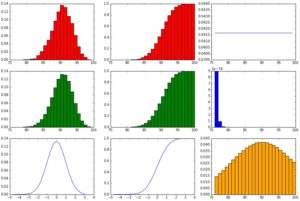
showHist(0.90, 100, 10000)
Nun ist die Wahrscheinlichkeit bei 90%. Der mögliche Ergebnisraum liegt zwar immer noch zwischen 0 und 100, aber es werden höhere Werte innerhalb dieses möglichen Ergebnisraumes wahrscheinlicher, niedrigere Werte unwahrscheinlicher.
Die Binominal-Verteilung und Normalverteilung sind der Erwartung entsprechend nach rechts verschoben, die Form hat sich aber nicht geändert (bei der zufällig generierten Binominal-Verteilung ändert sich die Form natürlich noch minimal durch die Zufallszahlen). Die Poisson-Verteilung zeigt eine überausgroße Varianz und gewinnt dermaßen an Breite, dass sie sich von der Binominal-Verteilung sehr weit entfernt hat. Die Geometrische Verteilung zeigt nun eine deutliche Reaktion in die andere Richtung, ihr Abfall ist sehr eindrucksvoll. Die Wahrscheinlichkeit, bereits nach dem ersten Durchgang gleich einen Erfolg zu haben, liegt schon bei 90%. Spätestens nach dem dritten Durchgang sollte der Erfolg eingetreten sein, alles andere wird äußerst unwahrscheinlich 🙂
Buchempfehlungen
Folgende zwei Bücher dienten zum Einen als Inspiration für diese Darstellung der Wahrscheinlichkeitsverteilungen, zum Anderen stellen sie eine weiterführende Lektüre dar:
 Einführung in Data Science: Grundprinzipien der Datenanalyse mit Python |
 Statistik-Workshop für Programmierer |



Leave a Reply
Want to join the discussion?Feel free to contribute!